| |
DNA Barcoding
Camiel Doorenweerd
Naturalis Biodiversity Center
Het principe
Soorten kunnen op basis van morfologie op naam gebracht worden, of aan de hand van het DNA. Dit is handig bij lastige soortsgroepen, taxonomische vraagstukken en biedt nieuwe mogelijkheden voor determinatie van premature stadia of resten. Zo kan men tegenwoordig al aan de hand van een watermonster uit een vijver zeggen of er beschermde amfibieën in voor komen. Het stukje DNA dat voor dieren internationaal gebruikt wordt is een fragment van 650 baseparen (bp, bestaande uit A, C, G of T) en wordt de DNA barcode genoemd.
Een database
Het gekozen barcode fragment is in de praktijk effectief gebleken voor determinaties en wordt universeel voor alle dieren gebruikt. De betrouwbaarheid van deze determinaties hangt voor een groot deel af van de kwaliteit en beschikbaarheid van referentie-DNA barcodes. Hiervoor is de Barcoding of Life Data Systems website opgezet (www.boldsystems.org  ). Hier wordt door onderzoekers en specialisten wereldwijd referentie barcodes aan toegevoegd. Een referentie barcode bestaat ten minste uit: 1) de DNA barcode sequentie, 2) vindplaats & taxonomie gegevens, 3) referentie waar de voucher [voucher = bewijsexemplaar waarvan het DNA is afgenomen] in een collectie te vinden is, en 4) een foto. Vlinders worden al sinds 2003 veel gebarcodeerd en zijn waarschijnlijk de groep die op dit moment het meest compleet is. ). Hier wordt door onderzoekers en specialisten wereldwijd referentie barcodes aan toegevoegd. Een referentie barcode bestaat ten minste uit: 1) de DNA barcode sequentie, 2) vindplaats & taxonomie gegevens, 3) referentie waar de voucher [voucher = bewijsexemplaar waarvan het DNA is afgenomen] in een collectie te vinden is, en 4) een foto. Vlinders worden al sinds 2003 veel gebarcodeerd en zijn waarschijnlijk de groep die op dit moment het meest compleet is.
Resultaten
In de database staan DNA barcodes voor meer dan 75.000 soorten vlinders wereldwijd (cijfers november 2013) en dit getal groeit elke dag. Het totaal aantal soorten vlinders wordt geschat op 300.000. Hiervan heeft nog maar ongeveer de helft een wetenschappelijke naam, dus er is nog werk te doen. Tijdens het verzamelen van DNA barcodes worden regelmatig verschillen gevonden binnen soorten die aanleiding geven om de soorten morfologisch nog eens goed te bekijken. Een klassiek voorbeeld was bij een dagvlinder uit Costa Rica, Astraptes fulgerator , die bij nader inzien uit tien verschillende soorten bleek te bestaan (http://www.pnas.org/content/101/41/14812/F2.expansion 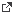 ). Morfologische verschillen waren onduidelijk in de adulten, maar wel aanwezig in de rupsen en waardplantkeuzes verschilden. Niet alleen in de tropen, maar ook in gebieden met een gematigd klimaat zijn er dergelijke gevallen. Bij een studie in 2013 naar de taxonomisch goed onderzochte Elachista bifasciella group (Elachistidae), bleek toch nog 34% meer soorten te zitten dan eerder gedacht (http://www.nature.com/srep/2013/131009/srep02901/full/srep02901.html ). Morfologische verschillen waren onduidelijk in de adulten, maar wel aanwezig in de rupsen en waardplantkeuzes verschilden. Niet alleen in de tropen, maar ook in gebieden met een gematigd klimaat zijn er dergelijke gevallen. Bij een studie in 2013 naar de taxonomisch goed onderzochte Elachista bifasciella group (Elachistidae), bleek toch nog 34% meer soorten te zitten dan eerder gedacht (http://www.nature.com/srep/2013/131009/srep02901/full/srep02901.html 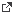 ). Het kan echter ook de andere kant op: DNA barcodes kunnen aanleiding geven tot synonymisering, zoals bijvoorbeeld bij Stigmella filipendulae en S. ulmariae (http://dare.uva.nl/cgi/arno/show.cgi?fid=363525 ). Het kan echter ook de andere kant op: DNA barcodes kunnen aanleiding geven tot synonymisering, zoals bijvoorbeeld bij Stigmella filipendulae en S. ulmariae (http://dare.uva.nl/cgi/arno/show.cgi?fid=363525 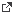 ). Zo zijn er nog tal van publicaties te noemen waar DNA barcodes een belangrijke rol hebben gespeeld, het wordt steeds meer een standaard methode. ). Zo zijn er nog tal van publicaties te noemen waar DNA barcodes een belangrijke rol hebben gespeeld, het wordt steeds meer een standaard methode.
Identificaties met DNA barcodes
Als er in de database voldoende referenties aanwezig zijn, kan een soort betrouwbaar aan de hand van het DNA op naam worden gebracht. Een voordeel hierbij is dat DNA in elke cel aanwezig is. Een afgevlogen vlinder is dus geen enkel probleem, rupsen hoeven niet altijd uitgekweekt te worden en zelfs minuscule resten kunnen soms gebruikt worden, een losse antenne bijvoorbeeld. Hoewel aanzienlijk goedkoper dan enkele jaren geleden, is het bepalen van een DNA barcode nog steeds redelijk kostbaar. Denk aan enkele tientjes per exemplaar. Er moet dus altijd gekeken worden naar alle mogelijkheden, blindelings alles barcoderen is geen optie.
Taxonomisch werk met DNA barcodes
Bij taxonomisch onderzoek moet DNA barcode data vooral gezien worden als een extra set kenmerken, zeker niet ter vervanging van morfologisch werk. Uiteindelijk moet naar het totaalplaatje van levenswijze, waardplant, morfologische kenmerken en DNA kenmerken gekeken worden om tot een taxonomische conclusie te komen. Een DNA barcode bestaat uit een combinatie van A, C, G en T, en wordt pas bruikbaar op het moment dat sequenties van verschillende exemplaren worden vergeleken. In de eerste plaats kan men dit doen door de sequenties onder elkaar te zetten, het zogenaamde 'alignen'. Daarmee zijn de verschillen echter niet duidelijk zichtbaar, dus een tweede stap is om de 'alignment' te vertalen naar een boom. De meest simpele statistische methode om een boom te maken is de 'neighbor-joining' methode. Hierin geven de horizontale taklengtes de genetische afstand aan tot de verschillende individuen (let op de schaalbalk). Dit lijkt ingewikkeld, maar als we bedenken dat de totale DNA barcode uit +/-650 baseparen bestaat, betekent een taklengte van 1% een verschil van 6,5 baseparen. Het genetische verschil tussen die twee exemplaren bestaat dan dus uit 6,5 mutaties. Door dit voor een groep exemplaren te doen, waarbij er zowel meerder individuen van één soort zijn als verschillende soorten, ontstaat een beeld van de genetische variatie binnen, en tussen, soorten. Hieruit kunnen conclusies worden getrokken over de afstand tussen soorten, zustersoortrelaties, of er (recente) voortplanting is enzovoorts.
Zelf DNA barcodes bepalen
Om een DNA barcode te bepalen is een moleculair laboratorium nodig. Er zijn in Nederland echter verschillende specialisten, onder andere bij Naturalis, die toegang hebben tot dergelijke labs en ook op de meeste universiteiten zijn de benodigde faciliteiten aanwezig. Het is vaak dus een kwestie van de juiste contacten leggen om DNA barcodes te verkrijgen. Bedenk wel dat DNA met de tijd degradeert. Een richtlijn is dat materiaal dat goed gedroogd is, zonder schimmels en dergelijke, tot 10 a 15 jaar oud nog gebruikt kan worden. Daarbij is niet het hele exemplaar nodig voor een analyse, een pootje of zelfs een antenne is voldoende. Ook kan het maken van een genitaalpreparaat gecombineerd worden met DNA extractie, dan is er geen enkel verlies van materiaal. Rupsen voor DNA onderzoek kunnen het beste in een ruim volume niet-gedenatureerde ethanol >95% bewaard worden, bladminerende rupsen kunnen eventueel met de mijn mee gedroogd worden.

Voorbeeld van een DNA barcode: 650 A, C, G en T's.

Voorbeeld van een Neighbor-joining boom met Perichares soorten (uit: Burns et al. 2008 http://www.pnas.org/content/105/17/6350.short ).
|